Project : Mesurer la contribution d’interactions individuelles entre miARN et cible sur un phénotype in vivo
Le miARN bantam a été découvert par un crible génétique chez la Drosophile : les homozygotes mutants meurent au stade pupal précoce, les hétérozygotes sont plus petits que les sauvages, et les mouches surexprimant bantam sont plus grosses que les sauvages ; les hypomorphes sont partiellement stériles femelles (Hipfner et al., 2002). La première cible proposée pour bantam a été le gène proapoptotique hid, dont la 3´ UTR contient plusieurs sites de complémentarité à la graine de bantam (Brennecke et al., 2003). De nombreuses autres cibles ont été proposées (les programmes de prédiction de cibles prédisent ≈ 70 cibles avec des sites de complémentarité conservée à bantam), certaines d’entre elles sont réprimées par bantam de manière mesurable au niveau de l’abondance de la protéine ou de l’ARNm, mais aucune validation in vivo rigoureuse de l’implication d’une cible dans le phénotype de croissance, de létalité ou de stérilité n’a encore été apportée. Par exemple, la régulation du gène enabled par bantam est très claire d’après des expériences de gène-rapporteur (Becam et al., 2011), mais l’ablation génétique des sites de reconnaissance de bantam sur le gène endogène enabled ne semble pas perturber le patron d’expression d’enabled ou déclencher un phénotype particulier (Bassett et al., 2014).
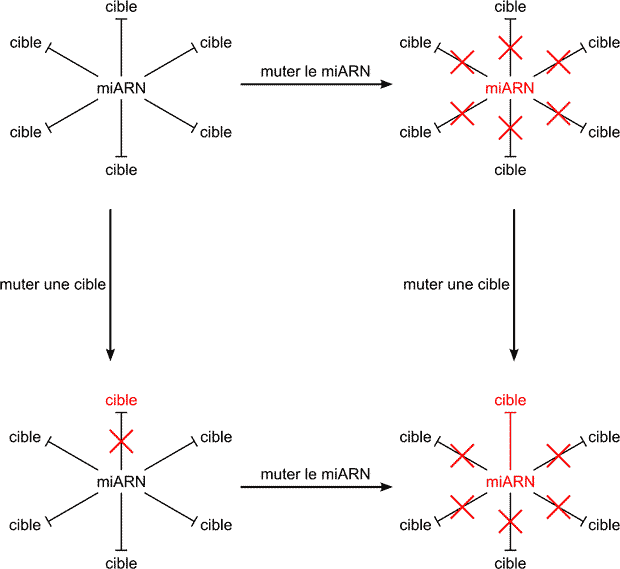
Nous nous concentrerons donc sur une stratégie purement in vivo pour mesurer la contribution de cibles individuelles au phénotype bantam. Par édition du génome, nous avons préparé des mouches mutantes dont la graine de bantam a été mutée en un autre hexamère. Nous sommes actuellement en train de préparer des mouches mutantes où les sites de reconnaissance de bantam dans le gène hid ont été mutées de manière compensatoire, afin d’isoler la contribution de l’interaction bantam/hid au phénotype global contrôlé par bantam (de façon similaire à la stratégie décrite par Ecsedi et al., 2015).