Projet : Régulation génique contre homéostasie : le cas des microARN
La régulation de l’expression génique, et l’homéostasie, sont deux principes universels du vivant. Chacun a été bien étudié, et les méthodes d’analyse modernes (biologie moléculaire à haut débit, bio-informatique, édition du génome) ont permis des avancées majeures dans leur compréhension. Mais elles n’ont jamais été vues sous une perspective commune, alors qu’elles présentent un antagonisme naturel : la régulation des gènes tend à modifier les réponses des systèmes biologiques, alors que les mécanismes homéostatiques leur donnent de l’inertie. Du dialogue entre ces deux propriétés du vivant, semblent émerger des caractéristiques inattendues.
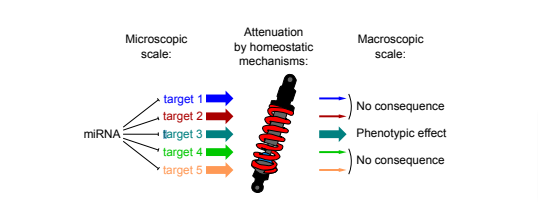
Le sujet de cette thèse concerne donc l’antagonisme entre l’homéostasie et un mode particulier de régulation des gènes : leur répression par les microARN. Ces ARN régulateurs ont la particularité de très peu réprimer leurs cibles (moins d’un facteur 2), tout en interagissant avec de très nombreuses cibles (quelques centaines de cibles par microARN chez les Mammifères). Les méthodes classiques d’identification des cibles, basées sur la biologie moléculaire et la bio-informatique, tendent à négliger cet aspect quantitatif. Le projet de thèse consiste à mesurer l’ampleur de l’atténuation homéostatique, et de la régulation par les microARN, sur un système simplifié (des cellules en culture), de manière à identifier les cibles dont la régulation survit à l’atténuation homéostatique, et est transmise de l’échelle microscopique à l’échelle mésoscopique. Les interactions ainsi identifiées seront ensuite testées in vivo chez la Drosophile, pour évaluer leur transmission jusqu’à l’échelle macroscopique, et de fait, identifier les interactions microARN/cibles responsables des phénotypes biologiques contrôlés par ces microARN.
Compétences mises en œuvre
- Biologie moléculaire (constructions plasmidiques, mesures d’abondance d’ARN, ...)
- Culture cellulaire (édition du génome, cytométrie, ...)
- Bio-informatique (recherche de motifs de séquence, analyses phylogénétiques, ...)
- Génétique in vivo (génétique de la Drosophile, notions de physiologie, ...)
Une maîtrise complète de ces techniques n’est pas requise en début de thèse (l’étudiant sera formé pendant la thèse), mais un intérêt certain pour la génétique moléculaire et les analyses mathématiques et informatiques des processus biologiques sera nécessaire.

